세그룹 이상의 평균을 비교하는 방법
김종엽
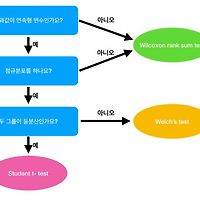
ANOVA라는 이름, 왠지 익숙하신가요? 학부 시절 커리큘럼에 통계 관련 과목이 하나만 있었어도 여러분은 틀림없이 이 단어를 배우셨을 겁니다. 활용 빈도 또한 무척 높죠. 사실 앞 챕터에서 알아봤던 두 그룹의 평균 비교는 논문을 작성할 때 실험군과 대조군의 나이 등을 비교하면서 자연스럽게 녹아들어가고, 실제로 결과 제출에 이용되는 분석 방법은 이 장에서 소개할 세 그룹 이상의 평균 비교부터일 겁니다. 앞장을 잘 이해하셨다면, 절대 어렵지 않습니다. 일단, 두 그룹의 비교에서처럼, 세 그룹 이상의 평균을 비교할 때 사용하는 통계분석방법도 3가지 뿐입니다. 그리고 고르는 방법도 아래 그림처럼 동일한 순서도를 가집니다. 앞장과 유사한 방법으로 진행하면 되겠죠?! 사실 그럴수도 있지만, 이번 챕터에서는 실제 연구에서 늘 부딪치게 되는 데이터 클리닝 과정을 살짝 엿보려고 합니다.
데이터 클리닝(본격적인 분석에 앞서 데이터 정리하기)
이번 챕터에서 이용할 예제 데이터는 건강보험심사평가원에서 공공데이터로 배포했던 대장암 환자 자료 중 300명을 임의로 추출한 것입니다. 데이터 안에는 환자의 나이(age), 성별(sex), 신장(height), 체중(weight), 대장암 병기(stage), 입원일수(HDday), 진단코드(Code) 등이 담겨 있습니다. 먼저 앞에서 배웠던 read.csv()함수를 이용해서 예제 데이터를 불러오도록 하겠습니다.
> colon300 <- read.csv(file = "colon300.csv", header = TRUE) # 'header = TRUE'라는
옵션은 예제데이터의 첫 번째 행이 각 열의 이름이라는 걸 컴퓨터에게 알려줍니다.
> #View(colon300) # 불러들인 colon300.csv파일이 colon300 라는 변수에 잘 담겼는지,
새로운 창을 열어 colon300 안을 보여달라는 명령어입니다.새로운 탭이 열리면서 아래와 같은 화면이 보인다면 성공입니다. 만약 그렇지 않다면, 틀림없이 경로설정의 문제일 겁니다. 이렇게 자신하는 이유는 저도 이 실수를 수도없이 반복하기 때문이죠. working directory가 파일이 위치한 폴더로 설정이 되어 있는지 확인하시기 바랍니다. 아차! 하며 직접 해결하실 수 있는 분들은 아래 결과를 확인한 이후에 계속 진행하시면 되고요. 만약 경로설정에 대해 1도 모르겠다시는 분이라면, 챕터3 ’엑셀파일 csv로 변환해서 R로 불러오기’와 부록 ’파일 경로 마스터하기’를 참고하시기 바랍니다.
데이터를 로드한 이후에 가장 먼저할 일은 어떤 데이터가 로드되었는지 확인하는 일입니다.물론, View()함수를 이용해서 확인하셨지만, 그걸로는 부족합니다. 데이터가 어떤 구조로 구성되어 있는지를 살피는게 진짜죠. 이를테면 심층면접 같은 겁니다. 구조를 보는 함수는 structure라는 단어의 약자인 str()함수입니다. 다음 코드를 입력하시고 Ctr + return(or enter) 키조합을 누르세요.
> str(colon300)'data.frame': 300 obs. of 7 variables:
$ age : int 32 46 63 52 55 53 72 75 64 71 ...
$ sex : Factor w/ 2 levels "F","M": 1 1 2 2 2 1 2 1 1 1 ...
$ height: num 167 167 157 151 165 ...
$ weight: num 56.7 71.9 49.7 53 50.5 89 54.4 70 55.2 63.8 ...
$ stage : Factor w/ 4 levels "I","II","III",..: 3 3 3 1 1 2 1 1 1 3 ...
$ code : Factor w/ 11 levels "C180","C181",..: 6 7 10 11 7 6 11 9 11 11 ...
$ HDday : int 8 9 8 11 32 15 19 65 10 8 ...결과를 살펴보도록 하죠. age는 int 형식으로 38, 52, 62 등등의 값이 담겨있다는 의미입니다. int는 interger의 약자로 정수를 의미합니다. height와 weight도 같은 숫자이지만, 형식은 num인 걸 볼 수가 있습니다. num은 number의 약자로 숫자를 의미합니다. int와는 달리 소숫점 아래의 숫자가 담을 수 있습니다. sex와 stage, code는 Factor라고 표시되어 있는데요. 한글로는 ’요인’이라고 번역합니다. 처음에는 문자가 담기는 chr형식과 혼돈스러우실 겁니다. 둘다 문자가 담기니까요. 하지만, chr에는 개별 문자가 모두 고유명사처럼 쓰인다면, 개별의 의미를 가진다면, Factor에서 문자는 종류(level) 중의 하나를 의미합니다. 예를 들어, sex는 2개의 levels “F”와 “M”으로 구성되어 있다는 의미이고, stage는 4개의 요인으로 구성되어 있으며, 개별 행은 4개의 요인 중 하나를 가지게 되죠. code는 11개로 구성되어 있음을 알 수 있습니다.
추가설명 : Factor w/4 levels “I”는 이 행은 4개 요인 중 하나인 ’I’값을 가진다는 걸 의미합니다. 단순히 ’I’라는 이름이 아니라, 요인 중 하나라는 의미는 R프로그램 뿐 아니라, 모든 통계 관련 프로그램에서 굉장히 큰 의미를 가집니다. 그러니, str()함수를 통해 데이터셋의 구조를 볼 때 해당열이 Factor인지 아니면 chr인지 구별해서 보는 실력을 키우셔야 합니다.
결측값(비어 있는 값)을 제거하기
실습을 위해 제작한 데이터셋을 제외하고 결측값이 하나도 없는 데이터는 존재하지 않습니다. 세상일이 그렇게 단순하지 않으니까요. View(colon300) 명령어를 통해 열어놓은 탭을 클릭해서 raw 데이터를 살펴보시면, 11번 째 환자 데이터의 경우 height 항목에 NA라고 표시되어 있는 걸 보실 수 있습니다. NA(또는 na)는 컴퓨터에서 값이 비어 있다는 의미입니다. ’0’과는 또다른 의미죠. 그런데 이런 값들이 통계분석에 그대로 포함되면 현상을 왜곡시킬 수가 있습니다. 그래서 연구자들은 크게 2가지 방법을 많이 활용합니다. 하나는 그 주변 행의 값들을 가지고 유추해서 결측값을 채우는 방법입니다. 두 번째 방법은 행(환자)에 하나의 값이라도 결측값(누락된 결과값)이 있으면 아예 그 행(환자)을 전체데이터셋에서 삭제하는 방법입니다. 저는 두 번째 방법을 선호합니다. 그런데 전체 환자 수가 만 명 단위를 넘어서면 수작업으로 결측값이 포함된 행(환자)을 모두 선별한다는 건 매우 어려운 일입니다. 그래서 결측값이 포함된 모든 행(환자)을 자동으로 제거해주는 함수가 있습니다. 바로 na.omit()이라는 함수입니다. 해당 함수를 써서, colon300 데이터셋에서 결측값이 하나도 없는 환자만을 추려 colon_clean이라는 변수에 담아보도록 하겠습니다.
> colon_clean <- na.omit(colon300)
명령이 실행된 이후에 위처럼, 우측 상단에 colon_clean 변수가 생성되었다면 성공하셨습니다. 그 뒤편의 숫자를 보면, colon300에서는 300개였던 관찰치(obs)가 296으로 줄어들었군요. height 값이 누락된 환자가 4명이 있었던 모양입니다.
데이터 살펴보기(통계값을 구하기 전에 탐색적 그래프를 그려보기)
이제 데이터 클리닝이 완료되었으니, 통계 분석의 첫 단추를 채워보도록 하겠습니다. 저는 기본 통계(회귀분석 등의 고급통계 말고)에서는 되도록 그래프를 먼저 그려서 여러분의 직관을 통해 결과를 유추해보라고 권합니다. 정규분포를 하는지 shapiro.test를 시행하기 전에 앞서 데이터의 분포가 실제로 정규분포(종 모양)에 가까운지 눈으로 확인해보라는 말씀입니다. 이렇게 하면, 나중에 구하게 될 통계값에 대해 자신을 가질 수도 있고 실수를 했을 때 다시 돌아볼 수 있는 기회가 생깁니다. 예를 들어, 정규분포를 절대 하지 않을 것 같은 모양의 분포였는데, 통계값이 정규분포라고 나왔다면 그건 100% 계산을 잘못한 겁니다. 통계값이란 눈으로 확인하면 알 수 있는 직관적인 결과를 남에게 신뢰성 있게 전달하기 위해 만들어진 것입니다. 이야기 만으로 ’내가 보기에는 정규분포 같더라’라고 하면 상대방이 믿어야할지 말아야할지 고민스러울 수 있겠죠. 그럴 때 p값을 적어준다면 상대방이 어느 정도 정규분포에 가까운지 객관적으로 느낄 수 있습니다. 거꾸로 눈으로 의미가 보이지 않는 결과에서 통계값이 의미있게 나온다면 전후가 뒤바뀐 거죠. (물론, 고급 통계에 가면 모든 결과를 그래프로 직관화하기 어렵습니다. 마치 고전역학은 그림으로 설명할 수 있지만, 양자역학은 어려운 것처럼요.)
분포를 살펴볼 때 가장 많이 활용되는 것은 밀도그래프입니다. 밀도 그래프를 그리는 함수는 여러 개가 있는데, 이 챕터에서는 문건웅 교수님이 제작해서 배포한 moonBook이라는 패키지에 들어 있는 densityplot()함수를 이용해보겠습니다.
> #install.packages("moonBook") # install.packages()함수는 여러분의 컴퓨터에서 한 번만
실행시키시면 됩니다. 마치 배틀그라운드를 윈도우에 설치하는 것과 같습니다.
컴퓨터를 떴다 켤때마다 게임을 다시 설치 하지 않는 것처럼요.
> library(moonBook) # 컴퓨터를 재시작하거나, 프로그램을 종료했다가
다시 실행시켰을 때에는 library()함수만 다시 실행시키면 됩니다.
이건 게임을 켜기 위해 게임 실행아이콘을 클릭하는 과정이라고 보시면 됩니다.
> moonBook::densityplot(height~stage, data=colon_clean)
명령어의 첫 번째 줄은 moonBook이라는 패키지를 여러분의 컴퓨터에 설치하라는 명령어입니다. 인터넷에서 패키지를 다운받아서 자동으로 설치를 시작합니다. 때문에 당연히 컴퓨터에 인터넷이 연결되어 있어야하고, 설치하는데 조금 시간이 걸립니다. 그 다음 줄의 library(moonBook) 명령어는 설치된 moonBook 패키지를 실행시키라는 의미입니다. 이 명령어를 실행시킨 이후부터 moonBook패키지 안에 들어 있는 여러 함수들을 사용할 수 있게 됩니다. 가장 먼저 살펴볼 함수는 densityplot()함수 입니다. moonBook::densityplot()이라고 함수 앞에 패키지의 이름(moonBook::)을 붙여쓴 걸 볼 수 있습니다. 이건 moonBook패키지 안에 있는 densityplot()함수를 지정하기 위해서입니다. 대부분은 패키지의 이름을 생략하고 쓰는데요. 여기에서만 패키지의 이름을 붙여준 건 densityplo()함수가 moonBook패키지 이외에 다른 패키지에도 동일한 이름의 함수가 들어 있기 때문입니다.
그래프를 보시면 다행히 네 그룹의 분포가 모두 종모양과 비슷한 걸 확인할 수 있습니다. 각각이 모두 정규분포를 할 가능성이 높겠군요. 그럼 shapiro.test를 통해 각 병기환자의 키 분포가 실제로도 정규분포를 하는지 살펴보겠습니다.
> shapiro.test(colon_clean$height[colon_clean$stage=="I"])
Shapiro-Wilk normality test
data: colon_clean$height[colon_clean$stage == "I"]
W = 0.97765, p-value = 0.3484> shapiro.test(colon_clean$height[colon_clean$stage=="II"])
Shapiro-Wilk normality test
data: colon_clean$height[colon_clean$stage == "II"]
W = 0.97606, p-value = 0.0819> shapiro.test(colon_clean$height[colon_clean$stage=="III"])
Shapiro-Wilk normality test
data: colon_clean$height[colon_clean$stage == "III"]
W = 0.98557, p-value = 0.2674> shapiro.test(colon_clean$height[colon_clean$stage=="IV"])
Shapiro-Wilk normality test
data: colon_clean$height[colon_clean$stage == "IV"]
W = 0.97757, p-value = 0.758
p-value가 0.3484, 0.819, 0.2674, 0.758로 모두 0.05이상인 걸 확인할 있습니다. shapiro.test에서는 p-value가 0.05일 때 정규분포를 한다고 해석합니다. 병기별 환자수를 살펴보는 명령어는 다음과 같습니다.
> table(colon_clean$stage)
I II III IV
59 94 113 30 각 병기별로 30명의 환자가 넘으니 각각의 분포가 정규분포를 하는 게 어쩌면 당연하겠군요. 그래서 우리는 일반적으로 30명이 넘으면 정규분포를 한다고 가정합니다. 집단의 분포를 알 수 없을 때 그룹별로 30명 이상씩의 대상자를 모집하는 이유도 동일합니다. 하지만 간과하면 안되는 게 있죠. 제가 앞 챕터에서 각 그룹의 정규분포가 중요한 게 아니라, 잔차(그룹 간 차이)의 분포가 정규분포를 하는지가 중요하다고 말씀드렸습니다. 잔차의 정규분포는 아래 명령어로 알아봅니다. 두 그룹일 때는 lm()함수를 썼었는데, 이번에는 aov()함수를 사용하는 게 차이점입니다.
> out = aov(height~stage, data=colon_clean)
> shapiro.test(resid(out))
Shapiro-Wilk normality test
data: resid(out)
W = 0.99132, p-value = 0.07844
결과의 해석은 p-value가 0.05보다 크다면 정규분포를 한다입니다. 0.07844로 넉넉하진 못하지만 정규분포를 가정할 수준은 되는군요. 제일 위의 순서도를 따라 내려가보록 하겠습니다. 정규분포를 하니까, 이번에는 등분산인지를 확인해야겠군요. 등분산을 확인하는 명령어는 다음과 같습니다.
> bartlett.test(height~stage, data=colon_clean) # p값이 0.05보다 크다면 등분산
Bartlett test of homogeneity of variances
data: height by stage
Bartlett's K-squared = 2.4437, df = 3, p-value = 0.4856해석은 정규분포 때와 동일하게 p-value가 0.05보다 크다면 등분산을 만족한다라고 읽으시면 됩니다. height값은 연속형 변수였고, 정규분포를 하며, 등분산도 만족하니, ANOVA를 통해 각 그룹의 평균을 비교하면 되겠군요.
ANOVA를 통한 그룹간의 평균 비교
이름도 유명한 ANOVA를 이용하는 방법은 의외로 간단합니다. 눈치 빠른 분들은 이미 알아채셨을텐데요. 정규분포를 확인하기 위해 out이라는 변수에 담아 두었던 값을 그대로 summary()함수에 담기만 하면 됩니다.
> out = aov(height~stage, data=colon_clean)
> summary(out) # p < 0.05이면 그룹간의 평균이 서로 다르다. Df Sum Sq Mean Sq F value Pr(>F)
stage 3 64 21.41 0.246 0.864
Residuals 292 25371 86.89 p-value가 0.864가 나왔네요. 우리가 통상적으로 차이가 있다는 가설을 검정하기 위해서는 p값이 0.05보다 작아야했었죠. p값이 0.05라는 의미는 이와 같은 분포가 자연적으로 발생할 확률이 5%라는 겁니다. 여기서는 0.864즉 차이가 없을 확률이 86.4%나 되니, 그룹간의 차이는 없다고 보는 게 옳겠죠?!
Kruskal Wallis H Test를 이용한 그룹간의 평균 비교
이번 예제데이터에서는 ANOVA를 써야했지만, 1) 결과값이 연속형 변수가 아니거나, 2) 연속형 변수였지만 정규분포를 하지 않는다면, Kruskal Wallis H Test로 평균을 비교해야합니다. 하는 방법은 아래와 같습니다.
> kruskal.test(height~stage, data=colon_clean) # p < 0.05이면 그룹간의 평균이 서로 같지 않다.
Kruskal-Wallis rank sum test
data: height by stage
Kruskal-Wallis chi-squared = 0.55222, df = 3, p-value = 0.9073해석은 ANOVA와 동일합니다.
Welch’s Test를 이용한 그룹간의 평균 비교
결과값이 연속형 변수였고, 정규분포는 하였으나, 등분산을 만족하지 않는 경우에는 Welch’s Test를 이용하게 됩니다. 하는 방법은 아래와 같습니다.
> oneway.test(height~stage, data=colon_clean, var.equal = FALSE) # p < 0.05이면
그룹간의 평균이 서로 같지 않다.
One-way analysis of means (not assuming equal variances)
data: height and stage
F = 0.26117, num df = 3.00, denom df = 104.79, p-value = 0.8532명령어 안에 var.equal = FALSE 라는 옵션을 지정해준 걸 볼 수 있습니다. var.equal은 등분산을 의미하고 TRUE가 아닌 FALSE는 만족하지 못했음을 의미합니다.
이렇게 여러 그룹의 평균을 비교하는 방법을 알아봤습니다. 보통 다른 통계책에서는 3 그룹의 평균을 비교하는 방법을 보여드리는데, 3 그룹 이상 즉 4 그룹이나 5 그룹의 평균비교도 동일하다는 점을 보여드리기 위해 저는 4 그룹으로 구성된 예제 데이터를 활용했습니다. 하지만 여기서 끝난 게 아니죠. 여러 그룹의 평균이 차이가 나는지는 알아봤지만, 만약 차이가 난다면 어떤 그룹과 어떤 그룹이 차이가 나서 그런 결과가 나왔는지는 아직 알아보지 않았으니까요. 다음 챕터에서는 바로 그 부분을 알아보도록 하겠습니다.
'깜신의 통계 이야기' 카테고리의 다른 글
| 2019년1월30일 통계워크샵 강의자료 (0) | 2019.01.29 |
|---|---|
| 2018년12월19일 통계워크샵 강의 자료 (1) | 2018.12.19 |
| 두 그룹의 평균을 비교하기 (2) | 2018.11.19 |
| 엑셀파일 CSV로 변환해서 R로 불러오기 (2) | 2018.10.26 |
| 생존분석(LogRankTest와CoxRegressionTest) - 깜신의 통계 왕초보 탈출 42탄 (4) | 2018.09.27 |
 colon300.csv
colon300.csv